数据案例展示
构建一个包含靶向人全基因组的sgRNA文库,文库包含63950 条sgRNA序列。文库构建完成后Sanger及NGS检测文库错误率,覆盖度及均一性。
文库错误率低
选取一个批次构建完成后随机抽取30个克隆进行桑格测序。测序结果表明在随机挑选的克隆中,100%的sgRNA序列和理论序列完全一致。
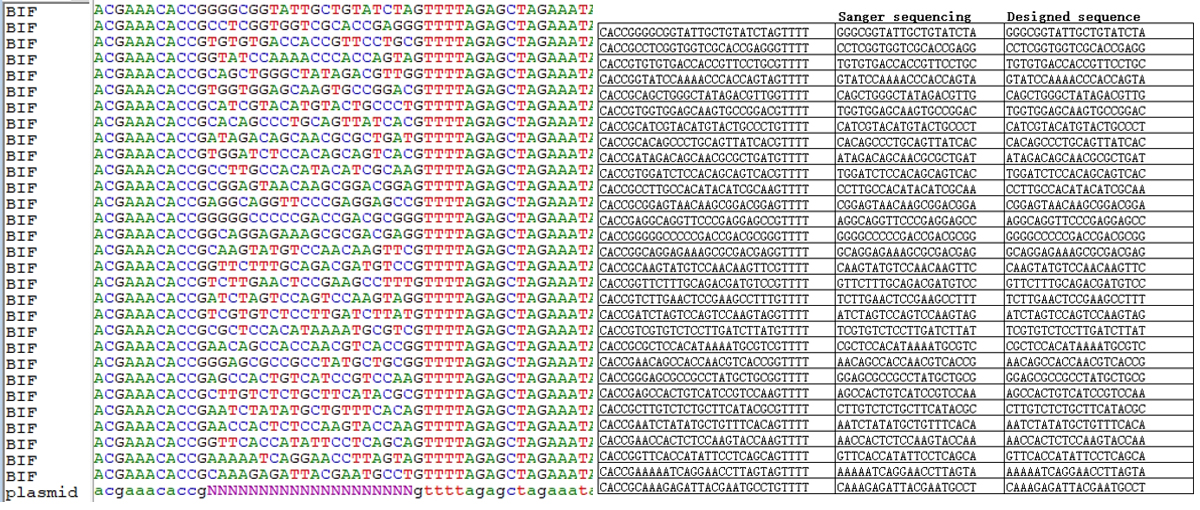
图1. 桑格测序结果
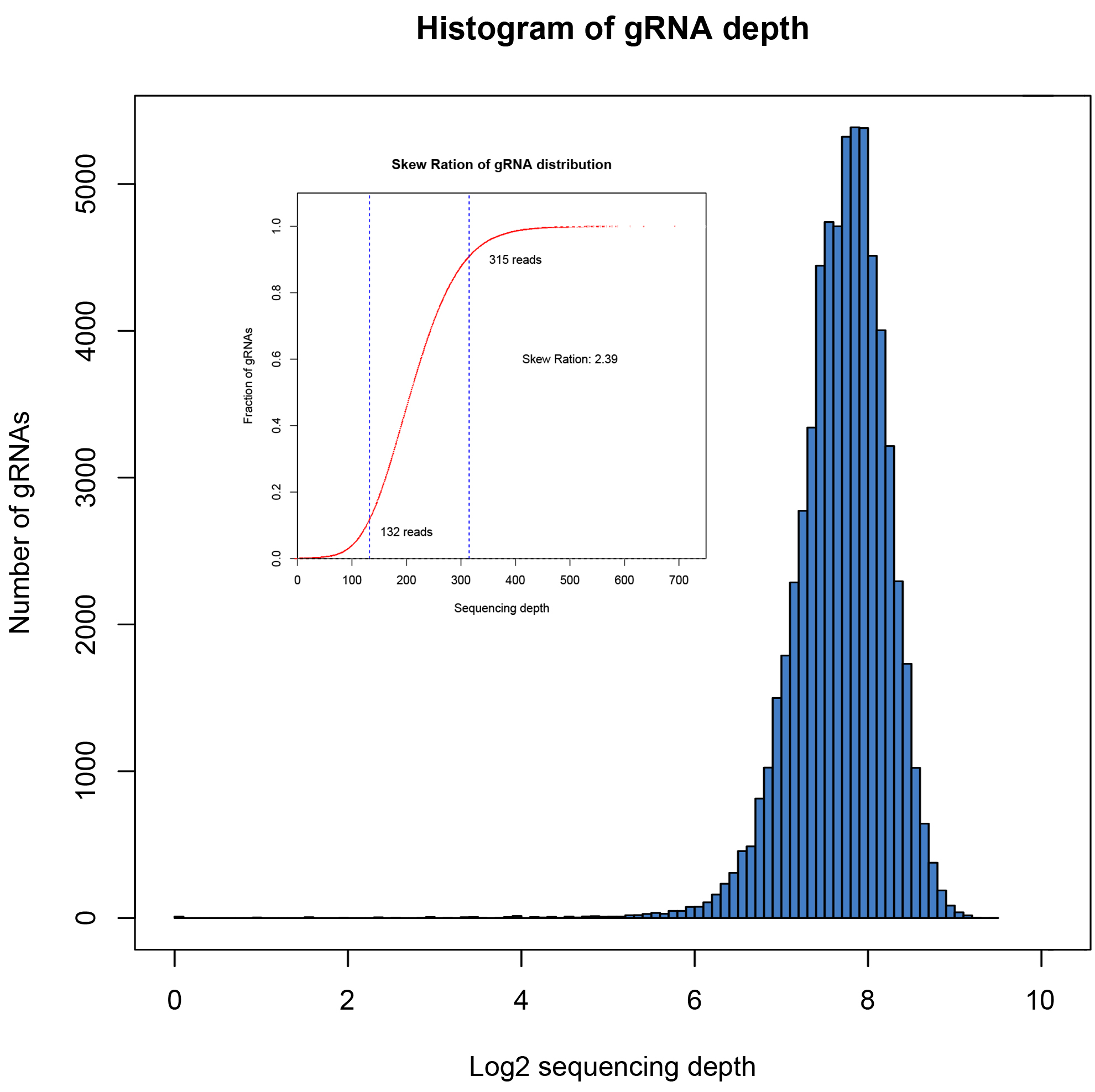
覆盖率高、分布均一,批次间稳定性高
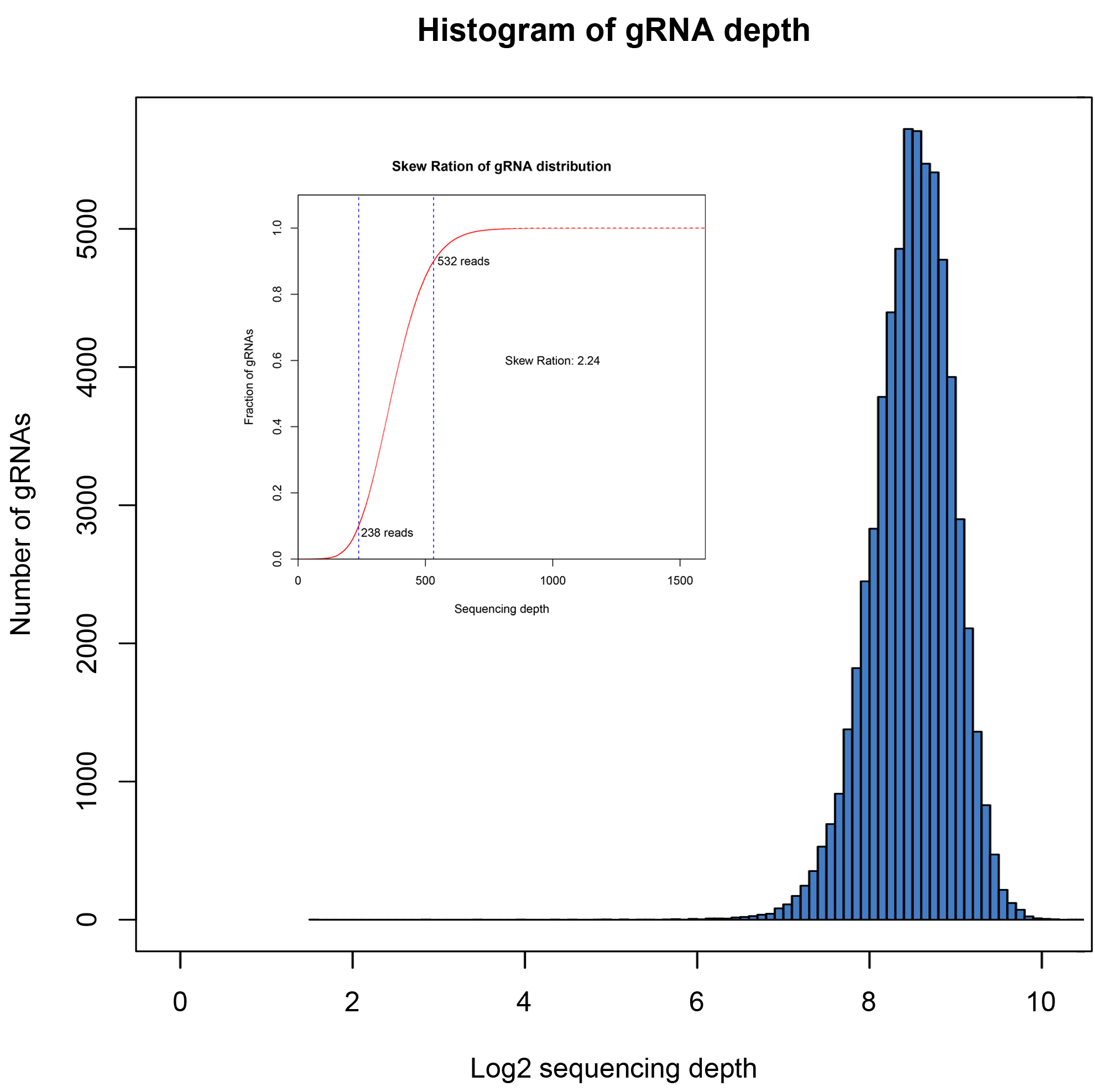
文库的覆盖度和分布均一性是评价文库质量的重要指标,使用NGS方法进行验证。覆盖率是指构建文库中包含目标序列的比例。理想状态下文库中不同sgRNA是均匀分布的,但因PCR过程及克隆过程会有一定的偏好性,导致一部分sgRNA reads数较低,一部分sgRNA reads数较高,因此使用均一性评价文库中序列分布的均匀程度,通常采用分布斜率Skew Ratio评价,通常认为数值<10代表为合格文库,数值越小代表均一性越好。
四个批次构建文库数据分析结果如下:
| 批次 |
理论gRNA多样性 |
实际gRNA多样性 |
覆盖度
(实际sgRNA多样性/理论多样性)
|
Skew Ratio |
| 01 |
63950 |
63950 |
100% |
2.24 |
| 02 |
63950 |
63940 |
99.98% |
2.39 |
| 03 |
63950 |
63937 |
99.98% |
2.91 |
| 04 |
63950 |
63943 |
99.99% |
2.29 |
 NGS杂交捕获DNA探针 QuarStar 94基因泛肿瘤Panel 3.0 QuarStar 176基因泛肿瘤Panel 3.0 QuarStar 227基因融合Panel 1.0 QuarStar 515基因泛肿瘤Panel 1.0 杂交捕获RNA探针 人全外显子组探针3.0 HRD panel 建库试剂 DNA建库试剂盒 片段化试剂 磁珠法捕获mRNA试剂盒 rRNA去除试剂盒 QuarPro T4连接酶 杂交捕获试剂 DNA探针快速杂交捕获试剂 DNA探针杂交捕获试剂V2版 DNA探针杂交捕获试剂 RNA探针一管式过夜杂交试剂 RNA探针快速杂交捕获试剂 接头体系 封闭液系统 扩增子NGS BRCA超多重引物 超多重PCR试剂盒2.0 PathoSeq 450病原库 配套试剂 链霉亲和素磁珠 设备与软件 iQuars 50自动化工作站
NGS杂交捕获DNA探针 QuarStar 94基因泛肿瘤Panel 3.0 QuarStar 176基因泛肿瘤Panel 3.0 QuarStar 227基因融合Panel 1.0 QuarStar 515基因泛肿瘤Panel 1.0 杂交捕获RNA探针 人全外显子组探针3.0 HRD panel 建库试剂 DNA建库试剂盒 片段化试剂 磁珠法捕获mRNA试剂盒 rRNA去除试剂盒 QuarPro T4连接酶 杂交捕获试剂 DNA探针快速杂交捕获试剂 DNA探针杂交捕获试剂V2版 DNA探针杂交捕获试剂 RNA探针一管式过夜杂交试剂 RNA探针快速杂交捕获试剂 接头体系 封闭液系统 扩增子NGS BRCA超多重引物 超多重PCR试剂盒2.0 PathoSeq 450病原库 配套试剂 链霉亲和素磁珠 设备与软件 iQuars 50自动化工作站 RNA合成sgRNA miRNA siRNA
RNA合成sgRNA miRNA siRNA



 引物与探针
引物与探针 基因合成
基因合成 寡核苷酸池
寡核苷酸池 CRISPR sgRNA定制文库
CRISPR sgRNA定制文库 抗体库
抗体库 突变体库
突变体库






 电话:400-017-9077
电话:400-017-9077 地址:上海市闵行区光华路248号5号楼2楼
地址:上海市闵行区光华路248号5号楼2楼 邮箱:
邮箱:







